爱湛蓝色的海洋
2021-06-09 02:31
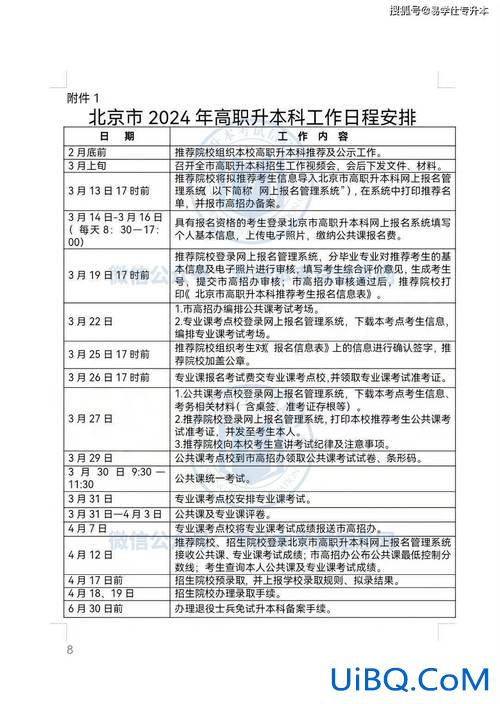
目前用于转录组数据获得和分析的方法主要有基于杂交技术的芯片技术,基于序列分析的基因表达系列分析(SAGE)和大规模平行信号测序系统(MPSS),以及最新提出的RNA-Seq技术等。
基因芯片是开发最早也是目前应用较广的高通量转录组检测技术。该技术成本适中,数据分析软件较多,整个方法较为成熟,然而基于杂交技术的微阵列技术只限用于已知序列,无法检测新的RNA;而且杂交技优艾设计网_设计百科术灵敏度有限,难以检测低丰度的目标(需要更多的样品量)和重复序列;也很难检测出融合基因转录、多顺反子转录等异常转录产物。与芯片不同,SAGE不需任何基因序列的信息,能够全局性地检测所有基因的表达水平,除了具有显示基因差异表达谱的作用外,还对那些未知基因特别是那些低拷贝基因的发现起到了巨大的推动作用。MPSS技术是对SAGE技术的改进,简化了测序过程,提高了精度,但二者都是基于昂贵的Sanger测序,需要大量的测序工作,技术难度较大,而且涉及酶切、PCR扩增、克隆等可能会产生碱基偏向性的操作步骤,因而限制了其推广。
相比之下,RNA-Seq技术具有诸多独特优势。(1)数字化信号:直接测定每个转录本片段序列,单核苷酸分辨率的精确度,可以检测单个碱基差异、基因家族中相似基因以及可变剪接造成的不同转录本的表达,同时不存在传统微阵列杂交的荧光模拟信号带来的交叉反应和背景噪音问题,能覆盖信号超高的动态变化范围。(2)高灵敏度:能够检测到细胞中少至几个拷贝的稀有转录本。(3)任意物种的全基因组分析:无需预先设计特异性探针,因此无需了解物种基因信息,能够直接对任何物种进行转录组分析,这对非模式生物的研究尤为重要,例如Wang等、Xiang等和Vera等利用RNA-Seq技术分别对白粉虱、海鲈鱼和蝴蝶转录组进行了研究。同时能够检测未知基因,发现新的转录本,并精确地识别可变剪切位点及cSNP,UTR区域。(4)更广的检测范围:高于6个数量级的动态检测范围,能够同时鉴定和定量稀有转录本和正常转录本;而芯片对过低或过高表达的基因缺乏敏感性,因而动态检测范围小。此外,RNA-Seq重复性好,无需技术重复,而且起始样品比芯片技术要少得多,尤其适用于来源极为有限的生物样品分析,如癌症干细胞。
目前用于转录组数据获得和分析的方法主要有基于杂交技术的芯片技术,基于序列分析的基因表达系列分析(SAGE)和大规模平行信号测序系统(MPSS),以及最新提出的RNA-Seq技术等。
基因芯片是开发最早也是目前应用较广的高通量转录组检测技术。该技术成本适中,数据分析软件较多,整个方法较为成熟,然而基于杂交技术的微阵列技术只限用于已知序列,无法检测新的RNA;而且杂交技优艾设计网_设计百科术灵敏度有限,难以检测低丰度的目标(需要更多的样品量)和重复序列;也很难检测出融合基因转录、多顺反子转录等异常转录产物。与芯片不同,SAGE不需任何基因序列的信息,能够全局性地检测所有基因的表达水平,除了具有显示基因差异表达谱的作用外,还对那些未知基因特别是那些低拷贝基因的发现起到了巨大的推动作用。MPSS技术是对SAGE技术的改进,简化了测序过程,提高了精度,但二者都是基于昂贵的Sanger测序,需要大量的测序工作,技术难度较大,而且涉及酶切、PCR扩增、克隆等可能会产生碱基偏向性的操作步骤,因而限制了其推广。
相比之下,RNA-Seq技术具有诸多独特优势。(1)数字化信号:直接测定每个转录本片段序列,单核苷酸分辨率的精确度,可以检测单个碱基差异、基因家族中相似基因以及可变剪接造成的不同转录本的表达,同时不存在传统微阵列杂交的荧光模拟信号带来的交叉反应和背景噪音问题,能覆盖信号超高的动态变化范围。(2)高灵敏度:能够检测到细胞中少至几个拷贝的稀有转录本。(3)任意物种的全基因组分析:无需预先设计特异性探针,因此无需了解物种基因信息,能够直接对任何物种进行转录组分析,这对非模式生物的研究尤为重要,例如Wang等、Xiang等和Vera等利用RNA-Seq技术分别对白粉虱、海鲈鱼和蝴蝶转录组进行了研究。同时能够检测未知基因,发现新的转录本,并精确地识别可变剪切位点及cSNP,UTR区域。(4)更广的检测范围:高于6个数量级的动态检测范围,能够同时鉴定和定量稀有转录本和正常转录本;而芯片对过低或过高表达的基因缺乏敏感性,因而动态检测范围小。此外,RNA-Seq重复性好,无需技术重复,而且起始样品比芯片技术要少得多,尤其适用于来源极为有限的生物样品分析,如癌症干细胞。







 加载中,请稍侯......
加载中,请稍侯......
精彩评论